Análise Correspondência Canônica
Adrielle Leal
2 de dezembro de 2021
Slides
Análise Multivariada – Correlação/Correspondência Canônica (CCA)
Agora que vocês já sabem que a Ordenação Canônica associa dois ou mais conjuntos de dados no próprio processo de ordenação, vocês serão capazes de extrair estruturas de um conjunto de dados (variável resposta) que estão relacionadas a outros conjuntos de dados (variável explicativa) e/ou testar hipóteses estatísticas sobre a significância dessas relações. A forma de combinar as informações de duas (ou, em alguns casos, mais) matrizes de dados depende do método de análise e nesta disciplina vamos aprender a análise de correspondência canônica (CCA).
Vamos colocar as mãos na massa?
Exemplo
O objetivo desse exemplo é desenvolver uma CCA da matriz de distâncias pelo método qui-quadrado de um conjunto de dados de composição de espécies de peixes “spe” para entender qual a relação com o conjunto de dados de variáveis ambientais “env2”.
Para isso, vamos usar a matriz de espécies de peixes e a matriz ambiental que vocês já trabalharam anteriormente. Em seguida, vamos desenvolver uma CCA através da função cca() do pacote vegan. Além disso, vamos plotar uma CCA e avaliar como o conjunto de dados de espécies se corresponde com os da matriz ambiental através da CCA e CA+envfit.
load("/home/felipe/Google Drive/github/eco_numerica/data/NEwR-2ed_code_data/NEwR2-Data/Doubs.RData")
spe## Cogo Satr Phph Babl Thth Teso Chna Pato Lele Sqce Baba Albi Gogo Eslu Pefl
## 1 0 3 0 0 0 0 0 0 0 0 0 0 0 0 0
## 2 0 5 4 3 0 0 0 0 0 0 0 0 0 0 0
## 3 0 5 5 5 0 0 0 0 0 0 0 0 0 1 0
## 4 0 4 5 5 0 0 0 0 0 1 0 0 1 2 2
## 5 0 2 3 2 0 0 0 0 5 2 0 0 2 4 4
## 6 0 3 4 5 0 0 0 0 1 2 0 0 1 1 1
## 7 0 5 4 5 0 0 0 0 1 1 0 0 0 0 0
## 8 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## 9 0 0 1 3 0 0 0 0 0 5 0 0 0 0 0
## 10 0 1 4 4 0 0 0 0 2 2 0 0 1 0 0
## 11 1 3 4 1 1 0 0 0 0 1 0 0 0 0 0
## 12 2 5 4 4 2 0 0 0 0 1 0 0 0 0 0
## 13 2 5 5 2 3 2 0 0 0 0 0 0 0 0 0
## 14 3 5 5 4 4 3 0 0 0 1 1 0 1 1 0
## 15 3 4 4 5 2 4 0 0 3 3 2 0 2 0 0
## 16 2 3 3 5 0 5 0 4 5 2 2 1 2 1 1
## 17 1 2 4 4 1 2 1 4 3 2 3 4 1 1 2
## 18 1 1 3 3 1 1 1 3 2 3 3 3 2 1 3
## 19 0 0 3 5 0 1 2 3 2 1 2 2 4 1 1
## 20 0 0 1 2 0 0 2 2 2 3 4 3 4 2 2
## 21 0 0 1 1 0 0 2 2 2 2 4 2 5 3 3
## 22 0 0 0 1 0 0 3 2 3 4 5 1 5 3 4
## 23 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0
## 24 0 0 0 0 0 0 1 0 0 2 0 0 1 0 0
## 25 0 0 0 0 0 0 0 0 1 1 0 0 2 1 0
## 26 0 0 0 1 0 0 1 0 1 2 2 1 3 2 1
## 27 0 0 0 1 0 0 1 1 2 3 4 1 4 4 1
## 28 0 0 0 1 0 0 1 1 2 4 3 1 4 3 2
## 29 0 1 1 1 1 1 2 2 3 4 5 3 5 5 4
## 30 0 0 0 0 0 0 1 2 3 3 3 5 5 4 5
## Rham Legi Scer Cyca Titi Abbr Icme Gyce Ruru Blbj Alal Anan
## 1 0 0 0 0 0 0 0 0 0 0 0 0
## 2 0 0 0 0 0 0 0 0 0 0 0 0
## 3 0 0 0 0 0 0 0 0 0 0 0 0
## 4 0 0 0 0 1 0 0 0 0 0 0 0
## 5 0 0 2 0 3 0 0 0 5 0 0 0
## 6 0 0 0 0 2 0 0 0 1 0 0 0
## 7 0 0 0 0 0 0 0 0 0 0 0 0
## 8 0 0 0 0 0 0 0 0 0 0 0 0
## 9 0 0 0 0 1 0 0 0 4 0 0 0
## 10 0 0 0 0 0 0 0 0 0 0 0 0
## 11 0 0 0 0 0 0 0 0 0 0 0 0
## 12 0 0 0 0 0 0 0 0 0 0 0 0
## 13 0 0 0 0 0 0 0 0 0 0 0 0
## 14 0 0 0 0 0 0 0 0 0 0 0 0
## 15 0 0 0 0 1 0 0 0 0 0 0 0
## 16 0 1 0 1 1 0 0 0 1 0 0 0
## 17 1 1 0 1 1 0 0 0 2 0 2 1
## 18 2 1 0 1 1 0 0 1 2 0 2 1
## 19 2 1 1 1 2 1 0 1 5 1 3 1
## 20 3 2 2 1 4 1 0 2 5 2 5 2
## 21 3 2 2 2 4 3 1 3 5 3 5 2
## 22 3 3 2 3 4 4 2 4 5 4 5 2
## 23 0 0 0 0 0 0 0 0 1 0 2 0
## 24 0 1 0 0 0 0 0 2 2 1 5 0
## 25 0 0 1 0 0 0 0 1 1 0 3 0
## 26 2 2 1 1 3 2 1 4 4 2 5 2
## 27 3 3 1 2 5 3 2 5 5 4 5 3
## 28 4 4 2 4 4 3 3 5 5 5 5 4
## 29 5 5 2 3 3 4 4 5 5 4 5 4
## 30 5 3 5 5 5 5 5 5 5 5 5 5spe[spe==0]<-1 ## filtrando os seus dados (ou seja, selecionando apenas linhas que contém 0) e substituindo por 1 na matriz de espécies
spe## Cogo Satr Phph Babl Thth Teso Chna Pato Lele Sqce Baba Albi Gogo Eslu Pefl
## 1 1 3 1 1 1 1 1 1 1 1 1 1 1 1 1
## 2 1 5 4 3 1 1 1 1 1 1 1 1 1 1 1
## 3 1 5 5 5 1 1 1 1 1 1 1 1 1 1 1
## 4 1 4 5 5 1 1 1 1 1 1 1 1 1 2 2
## 5 1 2 3 2 1 1 1 1 5 2 1 1 2 4 4
## 6 1 3 4 5 1 1 1 1 1 2 1 1 1 1 1
## 7 1 5 4 5 1 1 1 1 1 1 1 1 1 1 1
## 8 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## 9 1 1 1 3 1 1 1 1 1 5 1 1 1 1 1
## 10 1 1 4 4 1 1 1 1 2 2 1 1 1 1 1
## 11 1 3 4 1 1 1 1 1 1 1 1 1 1 1 1
## 12 2 5 4 4 2 1 1 1 1 1 1 1 1 1 1
## 13 2 5 5 2 3 2 1 1 1 1 1 1 1 1 1
## 14 3 5 5 4 4 3 1 1 1 1 1 1 1 1 1
## 15 3 4 4 5 2 4 1 1 3 3 2 1 2 1 1
## 16 2 3 3 5 1 5 1 4 5 2 2 1 2 1 1
## 17 1 2 4 4 1 2 1 4 3 2 3 4 1 1 2
## 18 1 1 3 3 1 1 1 3 2 3 3 3 2 1 3
## 19 1 1 3 5 1 1 2 3 2 1 2 2 4 1 1
## 20 1 1 1 2 1 1 2 2 2 3 4 3 4 2 2
## 21 1 1 1 1 1 1 2 2 2 2 4 2 5 3 3
## 22 1 1 1 1 1 1 3 2 3 4 5 1 5 3 4
## 23 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## 24 1 1 1 1 1 1 1 1 1 2 1 1 1 1 1
## 25 1 1 1 1 1 1 1 1 1 1 1 1 2 1 1
## 26 1 1 1 1 1 1 1 1 1 2 2 1 3 2 1
## 27 1 1 1 1 1 1 1 1 2 3 4 1 4 4 1
## 28 1 1 1 1 1 1 1 1 2 4 3 1 4 3 2
## 29 1 1 1 1 1 1 2 2 3 4 5 3 5 5 4
## 30 1 1 1 1 1 1 1 2 3 3 3 5 5 4 5
## Rham Legi Scer Cyca Titi Abbr Icme Gyce Ruru Blbj Alal Anan
## 1 1 1 1 1 1 1 1 1 1 1 1 1
## 2 1 1 1 1 1 1 1 1 1 1 1 1
## 3 1 1 1 1 1 1 1 1 1 1 1 1
## 4 1 1 1 1 1 1 1 1 1 1 1 1
## 5 1 1 2 1 3 1 1 1 5 1 1 1
## 6 1 1 1 1 2 1 1 1 1 1 1 1
## 7 1 1 1 1 1 1 1 1 1 1 1 1
## 8 1 1 1 1 1 1 1 1 1 1 1 1
## 9 1 1 1 1 1 1 1 1 4 1 1 1
## 10 1 1 1 1 1 1 1 1 1 1 1 1
## 11 1 1 1 1 1 1 1 1 1 1 1 1
## 12 1 1 1 1 1 1 1 1 1 1 1 1
## 13 1 1 1 1 1 1 1 1 1 1 1 1
## 14 1 1 1 1 1 1 1 1 1 1 1 1
## 15 1 1 1 1 1 1 1 1 1 1 1 1
## 16 1 1 1 1 1 1 1 1 1 1 1 1
## 17 1 1 1 1 1 1 1 1 2 1 2 1
## 18 2 1 1 1 1 1 1 1 2 1 2 1
## 19 2 1 1 1 2 1 1 1 5 1 3 1
## 20 3 2 2 1 4 1 1 2 5 2 5 2
## 21 3 2 2 2 4 3 1 3 5 3 5 2
## 22 3 3 2 3 4 4 2 4 5 4 5 2
## 23 1 1 1 1 1 1 1 1 1 1 2 1
## 24 1 1 1 1 1 1 1 2 2 1 5 1
## 25 1 1 1 1 1 1 1 1 1 1 3 1
## 26 2 2 1 1 3 2 1 4 4 2 5 2
## 27 3 3 1 2 5 3 2 5 5 4 5 3
## 28 4 4 2 4 4 3 3 5 5 5 5 4
## 29 5 5 2 3 3 4 4 5 5 4 5 4
## 30 5 3 5 5 5 5 5 5 5 5 5 5env## dfs ele slo dis pH har pho nit amm oxy bod
## 1 0.3 934 48.0 0.84 7.9 45 0.01 0.20 0.00 12.2 2.7
## 2 2.2 932 3.0 1.00 8.0 40 0.02 0.20 0.10 10.3 1.9
## 3 10.2 914 3.7 1.80 8.3 52 0.05 0.22 0.05 10.5 3.5
## 4 18.5 854 3.2 2.53 8.0 72 0.10 0.21 0.00 11.0 1.3
## 5 21.5 849 2.3 2.64 8.1 84 0.38 0.52 0.20 8.0 6.2
## 6 32.4 846 3.2 2.86 7.9 60 0.20 0.15 0.00 10.2 5.3
## 7 36.8 841 6.6 4.00 8.1 88 0.07 0.15 0.00 11.1 2.2
## 8 49.1 792 2.5 1.30 8.1 94 0.20 0.41 0.12 7.0 8.1
## 9 70.5 752 1.2 4.80 8.0 90 0.30 0.82 0.12 7.2 5.2
## 10 99.0 617 9.9 10.00 7.7 82 0.06 0.75 0.01 10.0 4.3
## 11 123.4 483 4.1 19.90 8.1 96 0.30 1.60 0.00 11.5 2.7
## 12 132.4 477 1.6 20.00 7.9 86 0.04 0.50 0.00 12.2 3.0
## 13 143.6 450 2.1 21.10 8.1 98 0.06 0.52 0.00 12.4 2.4
## 14 152.2 434 1.2 21.20 8.3 98 0.27 1.23 0.00 12.3 3.8
## 15 164.5 415 0.5 23.00 8.6 86 0.40 1.00 0.00 11.7 2.1
## 16 185.9 375 2.0 16.10 8.0 88 0.20 2.00 0.05 10.3 2.7
## 17 198.5 349 0.5 24.30 8.0 92 0.20 2.50 0.20 10.2 4.6
## 18 211.0 333 0.8 25.00 8.0 90 0.50 2.20 0.20 10.3 2.8
## 19 224.6 310 0.5 25.90 8.1 84 0.60 2.20 0.15 10.6 3.3
## 20 247.7 286 0.8 26.80 8.0 86 0.30 3.00 0.30 10.3 2.8
## 21 282.1 262 1.0 27.20 7.9 85 0.20 2.20 0.10 9.0 4.1
## 22 294.0 254 1.4 27.90 8.1 88 0.20 1.62 0.07 9.1 4.8
## 23 304.3 246 1.2 28.80 8.1 97 2.60 3.50 1.15 6.3 16.4
## 24 314.7 241 0.3 29.76 8.0 99 1.40 2.50 0.60 5.2 12.3
## 25 327.8 231 0.5 38.70 7.9 100 4.22 6.20 1.80 4.1 16.7
## 26 356.9 214 0.5 39.10 7.9 94 1.43 3.00 0.30 6.2 8.9
## 27 373.2 206 1.2 39.60 8.1 90 0.58 3.00 0.26 7.2 6.3
## 28 394.7 195 0.3 43.20 8.3 100 0.74 4.00 0.30 8.1 4.5
## 29 422.0 183 0.6 67.70 7.8 110 0.45 1.62 0.10 9.0 4.2
## 30 453.0 172 0.2 69.00 8.2 109 0.65 1.60 0.10 8.2 4.4env2<- env[,-1] ## removendo 'dfs' que é uma variável espacial e não ambientalAgora instale e/ou carregue os pacotes necessários:
library(permute)
library(lattice)
library(vegan)Faça uma CCA:
resultado.cca <- cca(spe, env2)
resultado.cca## Call: cca(X = spe, Y = env2)
##
## Inertia Proportion Rank
## Total 0.27495 1.00000
## Constrained 0.17528 0.63751 10
## Unconstrained 0.09966 0.36249 19
## Inertia is scaled Chi-square
##
## Eigenvalues for constrained axes:
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6 CCA7 CCA8 CCA9 CCA10
## 0.13097 0.01509 0.00966 0.00631 0.00549 0.00310 0.00198 0.00143 0.00084 0.00041
##
## Eigenvalues for unconstrained axes:
## CA1 CA2 CA3 CA4 CA5 CA6 CA7 CA8
## 0.026280 0.021516 0.011876 0.009520 0.006698 0.005848 0.004507 0.003578
## (Showing 8 of 19 unconstrained eigenvalues)summary(resultado.cca)##
## Call:
## cca(X = spe, Y = env2)
##
## Partitioning of scaled Chi-square:
## Inertia Proportion
## Total 0.27495 1.0000
## Constrained 0.17528 0.6375
## Unconstrained 0.09966 0.3625
##
## Eigenvalues, and their contribution to the scaled Chi-square
##
## Importance of components:
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## Eigenvalue 0.1310 0.01509 0.009656 0.00631 0.005489 0.003101
## Proportion Explained 0.4763 0.05490 0.035118 0.02295 0.019963 0.011280
## Cumulative Proportion 0.4763 0.53123 0.566344 0.58929 0.609256 0.620536
## CCA7 CCA8 CCA9 CCA10 CA1 CA2
## Eigenvalue 0.001984 0.001433 0.0008367 0.0004147 0.02628 0.02152
## Proportion Explained 0.007214 0.005212 0.0030432 0.0015084 0.09558 0.07826
## Cumulative Proportion 0.627750 0.632963 0.6360059 0.6375143 0.73310 0.81135
## CA3 CA4 CA5 CA6 CA7 CA8
## Eigenvalue 0.01188 0.00952 0.006698 0.005848 0.004507 0.003578
## Proportion Explained 0.04319 0.03462 0.024360 0.021269 0.016392 0.013013
## Cumulative Proportion 0.85455 0.88917 0.913529 0.934798 0.951190 0.964204
## CA9 CA10 CA11 CA12 CA13 CA14
## Eigenvalue 0.002456 0.002062 0.001883 0.001481 0.0006381 0.0004729
## Proportion Explained 0.008934 0.007501 0.006847 0.005386 0.0023206 0.0017198
## Cumulative Proportion 0.973138 0.980639 0.987486 0.992872 0.9951929 0.9969127
## CA15 CA16 CA17 CA18 CA19
## Eigenvalue 0.0003757 0.0001988 0.0001737 0.0000857 1.499e-05
## Proportion Explained 0.0013664 0.0007230 0.0006317 0.0003117 5.451e-05
## Cumulative Proportion 0.9982791 0.9990021 0.9996338 0.9999455 1.000e+00
##
## Accumulated constrained eigenvalues
## Importance of components:
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## Eigenvalue 0.1310 0.01509 0.009656 0.00631 0.005489 0.003101
## Proportion Explained 0.7472 0.08611 0.055086 0.03600 0.031314 0.017693
## Cumulative Proportion 0.7472 0.83328 0.888363 0.92436 0.955675 0.973368
## CCA7 CCA8 CCA9 CCA10
## Eigenvalue 0.001984 0.001433 0.0008367 0.0004147
## Proportion Explained 0.011316 0.008176 0.0047735 0.0023661
## Cumulative Proportion 0.984684 0.992860 0.9976339 1.0000000
##
## Scaling 2 for species and site scores
## * Species are scaled proportional to eigenvalues
## * Sites are unscaled: weighted dispersion equal on all dimensions
##
##
## Species scores
##
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## Cogo -0.39760 0.094136 -0.233691 -0.092110 0.021110 -0.005211
## Satr -0.75799 -0.158852 -0.010532 0.047333 0.019866 0.024018
## Phph -0.71180 -0.032540 0.058472 0.061413 0.028563 0.001592
## Babl -0.63018 0.034304 0.159897 0.032006 0.079079 0.011610
## Thth -0.44108 0.054855 -0.286798 -0.072331 -0.030538 0.012686
## Teso -0.37256 0.200054 -0.202466 -0.116425 0.046272 -0.112209
## Chna -0.09098 0.141681 0.001848 0.009471 -0.050826 0.120863
## Pato -0.09201 0.247401 0.032126 0.091081 -0.112171 -0.019470
## Lele -0.05325 0.097412 0.053922 -0.131477 -0.068882 -0.031691
## Sqce 0.11079 0.068912 0.117294 -0.177139 -0.003607 -0.005713
## Baba 0.19924 0.145336 0.023627 0.119639 -0.082822 -0.078456
## Albi 0.03235 -0.040080 -0.023703 0.114792 -0.197441 0.005776
## Gogo 0.25338 0.102453 -0.002716 0.081029 0.016913 -0.038877
## Eslu 0.18785 -0.135111 0.081389 -0.063229 -0.019895 0.052934
## Pefl 0.06784 -0.089254 0.067764 -0.086109 -0.162885 0.049538
## Rham 0.25651 -0.070222 -0.010586 0.088103 -0.038315 -0.056662
## Legi 0.22484 -0.093182 -0.018125 0.058021 0.006999 -0.010168
## Scer 0.05627 -0.137952 -0.029432 -0.084300 -0.062337 0.053107
## Cyca 0.18424 -0.153378 -0.067157 -0.037664 0.007655 -0.036116
## Titi 0.25127 0.009952 0.063891 0.022348 0.099877 -0.011972
## Abbr 0.24742 -0.102302 -0.045975 -0.006548 0.033795 0.011812
## Icme 0.15820 -0.253397 -0.086430 -0.016460 -0.041592 0.011711
## Gyce 0.38551 -0.057993 -0.034823 0.019233 0.144025 -0.011319
## Ruru 0.28077 0.083626 0.142548 -0.083443 0.037507 -0.020443
## Blbj 0.31023 -0.079776 -0.024391 0.021415 0.050653 -0.070912
## Alal 0.39890 0.175120 -0.088994 0.057737 0.067081 0.159171
## Anan 0.24385 -0.162673 -0.041751 0.028545 0.027950 -0.032589
##
##
## Site scores (weighted averages of species scores)
##
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## 1 -0.31964 -0.98051 -1.52084 -0.111515 -0.9061 0.23402
## 2 -1.29927 -1.42796 0.13892 1.419795 0.7052 0.86953
## 3 -1.58545 -1.25684 1.13274 1.820265 1.5232 1.00779
## 4 -1.35231 -1.33394 1.51787 0.995531 0.5621 1.61503
## 5 0.01380 -0.26068 2.70885 -4.299083 -2.0078 0.61443
## 6 -1.10677 -0.54175 1.55440 0.571648 1.6974 0.46313
## 7 -1.48414 -1.23318 1.00318 1.612036 1.4264 1.02081
## 8 0.08541 -0.27353 -1.55270 -0.675438 -1.2414 -0.32231
## 9 0.06938 0.89014 2.33549 -4.446103 0.3658 -0.78775
## 10 -0.79987 0.11386 1.24733 -0.649472 0.3460 -0.22836
## 11 -0.79921 -1.09071 -0.81054 0.811406 -0.3333 0.26021
## 12 -1.48691 -1.00673 -0.82934 0.772404 0.9763 0.96044
## 13 -1.50034 -0.72687 -2.75199 -0.005072 0.4333 -0.04018
## 14 -1.75301 -0.03119 -3.37408 -0.768242 1.1747 -0.61980
## 15 -1.31120 1.18352 -1.30963 -2.209727 0.8078 -2.83108
## 16 -1.06468 2.63146 -0.54395 -1.763944 -0.8969 -4.46003
## 17 -0.60511 1.73194 0.96443 1.297245 -4.0493 -1.01599
## 18 -0.10112 1.36121 1.26157 0.717463 -4.2467 -0.60098
## 19 -0.08401 2.29460 1.88293 1.792782 -0.1397 0.33208
## 20 0.81885 1.49759 0.81200 1.032279 -0.9280 0.73324
## 21 1.03111 0.70071 0.23991 0.838622 -0.2338 0.46859
## 22 1.06454 0.37091 0.37855 -0.350534 -0.1381 0.14226
## 23 0.19114 0.15061 -1.82642 -0.324517 -0.7605 1.52219
## 24 0.60067 1.33203 -1.63193 -0.584860 1.4055 5.42676
## 25 0.34441 0.75359 -2.02126 0.430188 -0.1997 2.71366
## 26 1.04195 0.62429 -0.49408 0.643017 3.1132 1.46718
## 27 1.22124 -0.19315 0.09362 0.515897 2.8276 -1.01433
## 28 1.22522 -1.18615 -0.36926 -0.212699 2.0657 -1.33422
## 29 1.14253 -0.96965 -0.02063 0.426195 -1.0103 -0.52849
## 30 1.14484 -2.04759 -0.54736 -0.001465 -1.3825 -0.30822
##
##
## Site constraints (linear combinations of constraining variables)
##
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## 1 -0.40031 -1.3402 -1.45775 0.24370 -0.29775 0.21975
## 2 -0.92944 -0.8674 2.46042 1.66262 0.42661 1.22778
## 3 -1.32491 -1.5223 0.71535 0.73998 1.48890 0.46962
## 4 -1.41575 -0.3413 1.53658 -0.19597 -0.24815 0.04155
## 5 -0.51261 -0.7459 1.04326 -1.96105 -0.16839 0.73071
## 6 -1.46462 -0.8715 0.70764 1.45141 2.00079 1.27303
## 7 -1.45774 -0.6939 0.43894 -1.42762 -0.88762 -0.42483
## 8 -0.09709 -0.5047 0.57935 -2.97417 0.14712 0.70677
## 9 0.15015 0.1175 1.89146 -2.59713 -0.23801 0.29528
## 10 -0.37088 0.4789 0.66069 -0.01611 -0.54520 0.61027
## 11 -1.12231 0.1031 -0.64202 0.02435 0.41632 -1.15191
## 12 -1.30481 0.3258 -0.44177 1.01187 -1.05469 0.36888
## 13 -1.34868 0.3329 -1.02225 -0.38599 -1.66268 -0.42344
## 14 -1.66396 -0.1474 -1.96452 0.08394 0.55950 -1.03939
## 15 -1.20828 0.1350 -1.52223 -0.91413 0.87971 -1.36100
## 16 -0.19564 1.5039 0.50041 -0.11972 -0.07193 -0.76404
## 17 -0.07334 0.3982 -0.07530 0.91978 -0.60256 -0.38577
## 18 -0.14284 1.1453 0.40878 0.22700 -0.84578 -0.44424
## 19 -0.44667 0.9987 -0.22716 0.82363 0.62890 -0.50499
## 20 0.33197 0.9857 0.66360 1.11559 -1.53279 -0.55361
## 21 0.74922 1.2930 0.78172 0.47937 -0.12214 -0.06240
## 22 0.57790 1.0693 -0.18529 -0.44650 -0.08940 -0.07244
## 23 -0.14353 0.5852 -3.01725 -0.05881 -0.06509 3.09754
## 24 1.27644 0.9802 -0.73684 -1.58590 0.03486 1.90077
## 25 0.46628 0.8537 -1.23958 0.55046 -0.56638 3.30234
## 26 0.94984 0.6448 -0.08766 0.20026 3.04738 0.54687
## 27 1.53871 0.2117 0.27759 0.10109 0.66936 -0.20219
## 28 1.16114 -0.2289 -0.01214 0.09162 0.92917 -1.70795
## 29 1.05208 -1.6606 -0.07161 1.06919 -1.31683 0.19034
## 30 1.25212 -1.8939 -0.67185 -0.36660 -0.22126 -0.45674
##
##
## Biplot scores for constraining variables
##
## CCA1 CCA2 CCA3 CCA4 CCA5 CCA6
## ele -0.7224 -0.274894 0.43672 -0.226500 0.08328 0.21349
## slo -0.1913 -0.202421 -0.11715 0.002381 -0.05504 0.06589
## dis 0.7410 -0.276467 -0.36062 0.232229 -0.12755 -0.16719
## pH -0.1684 -0.122439 -0.34199 -0.317932 0.31377 -0.48298
## har 0.5247 -0.003076 -0.43098 -0.227576 -0.26978 -0.20470
## pho 0.3281 0.155122 -0.42310 0.002059 0.14220 0.57510
## nit 0.6258 0.405410 -0.27214 0.218336 0.08816 0.05827
## amm 0.3049 0.239254 -0.30609 0.030636 -0.01058 0.62654
## oxy -0.7241 -0.038428 -0.03152 0.282902 -0.23950 -0.50782
## bod 0.3647 0.156648 -0.35400 -0.186430 0.19609 0.74024Os primeiros resultados mostram apenas os valores referentes ao conjunto de dados “spe”. Na sequência, “constrained” integra-se os resultados de “env2” nos valores apresentados para identificar o quanto a adição da matriz de “spe” auxilia na interpretação do conjunto total de resultados da análise de correlação canônica.
plot(resultado.cca)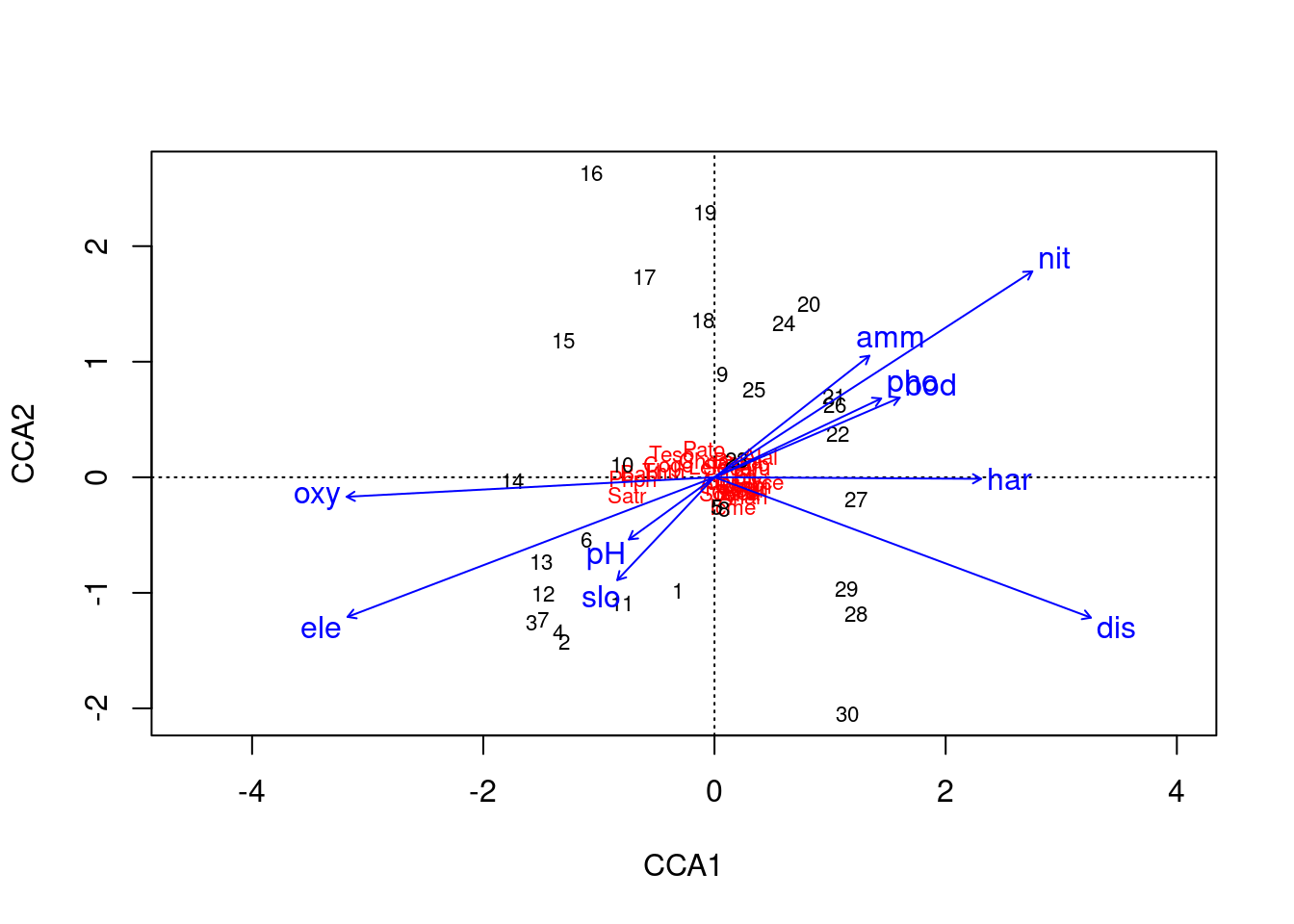
Comparação da CCA com CA + Envfit
resultado.ca<-cca(spe)
resultado.envfit<-envfit(resultado.ca, env2) #resultado de permutação de cada variável do env2
resultado.envfit##
## ***VECTORS
##
## CA1 CA2 r2 Pr(>r)
## ele -0.99119 0.13242 0.4567 0.001 ***
## slo -0.78974 0.61345 0.0498 0.583
## dis 0.97827 0.20736 0.4993 0.001 ***
## pH -0.74346 0.66878 0.0489 0.537
## har 0.99792 -0.06441 0.2355 0.027 *
## pho 0.99505 0.09940 0.0970 0.287
## nit 0.97174 -0.23606 0.3596 0.007 **
## amm 0.98576 -0.16815 0.0867 0.329
## oxy -0.99990 0.01383 0.4682 0.002 **
## bod 0.99487 0.10121 0.1224 0.198
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## Permutation: free
## Number of permutations: 999Comparação de Gráficos CCA com CA + Envfit
par(mfrow=c(1,2))
plot(resultado.cca, main="CCA")
plot(resultado.ca, main="CA + Envfit")
plot(resultado.envfit)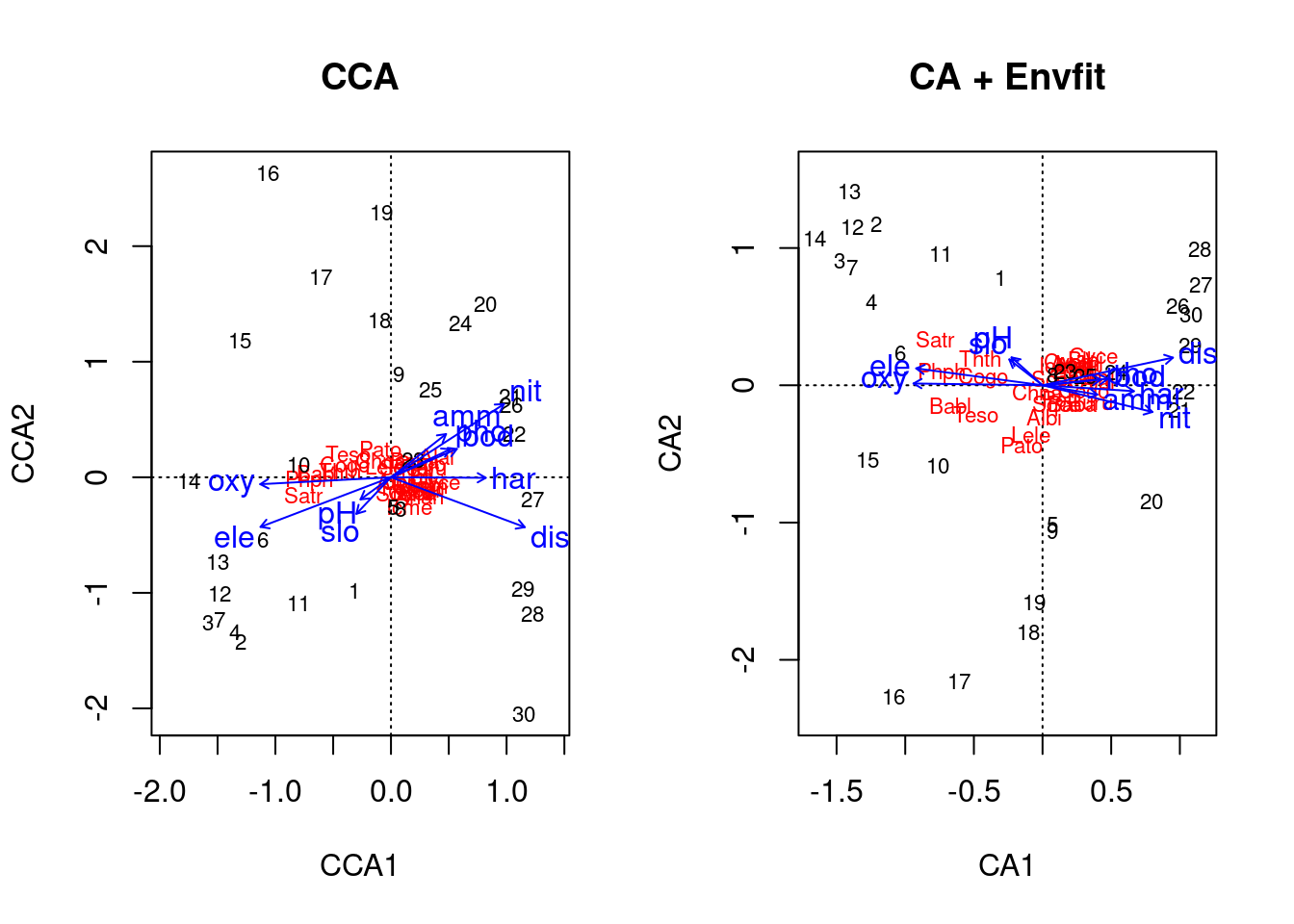
Existem diferenças na construção estatística dos gráficos, porém em termos conceituais são a mesma coisa.
Praticamente nada da interpretação dos resultados vai mudar. Na situação com envfit, temos o resultado de significância para cada variável ambiental.
Agora é com vocês!
Como vocês já sabem, a CCA é extremamente influenciada por espécies raras. Diante disso, vocês conseguem detectar algumas espécies raras na nossa matriz “spe”? Se sim, o que vocês devem fazer com isso para seguir a análise?
Agora que vocês resolveram essa questão, faça um CCA com a nova matriz “spe” e os dados ambientais. Plot esse resultado e discorra sobre o que foi encontrado.
Teste a significância global e de cada eixo gerado. Interprete o que você encontrou.